Машинное обучение в биологии: и на Nature бывает проруха
В январском номере Nature Reviews Molecular Cell Biology был опубликован обзор, посвящённый машинному обучению, под названием “A guide to machine learning for biologists”. Увлекательное название, неправда ли? Когда статья была опубликована, её прочёл биолог Дмитрий Пензар (младший научный сотрудник ИОГен РАН, преподаватель ФББ МГУ) активно использующий машинное обучение в своей работе и читающий курсы, посвящённые ему. По мнению ученого, обзор, опубликованный в таком крутом журнале оказался очень слабым и содержал много ошибок. Что же тогда читать и слушать, если нужен настоящий guide to machine learning for biologists? Мы решили поговорить с исследователем - о статье в журнале и о машинном обучении в биологии вообще.

Дмитрий, расскажите, пожалуйста, что вам не понравилось в этом обзоре. Нам и нашим читателям интересно узнать, как вдруг так оказалось, что обзор из Nature Reviews — посредственный.
Во-первых, характер статьи — вводный, в плохом смысле этого слова. Прочитав заголовок, ожидаешь от статьи информацию по современным методам, используемым в биологии, и интуицию, которая стоит за их использованием. Эта же статья представляет собой дикую смесь из вводных глав учебника по ML, воды и того, что можно нагуглить по указанным темам за 5 минут.
Естественно, это приводит к еще одной проблеме — жуткая невычитанность и внутренняя несвязность статьи. Например, авторы упоминают автоэнкодеры как средство решения проблемы уменьшения размерности, но в схеме, которую они приводят для того, чтобы показать когда что использовать, автоэнкодеры вовсе не упоминаются.
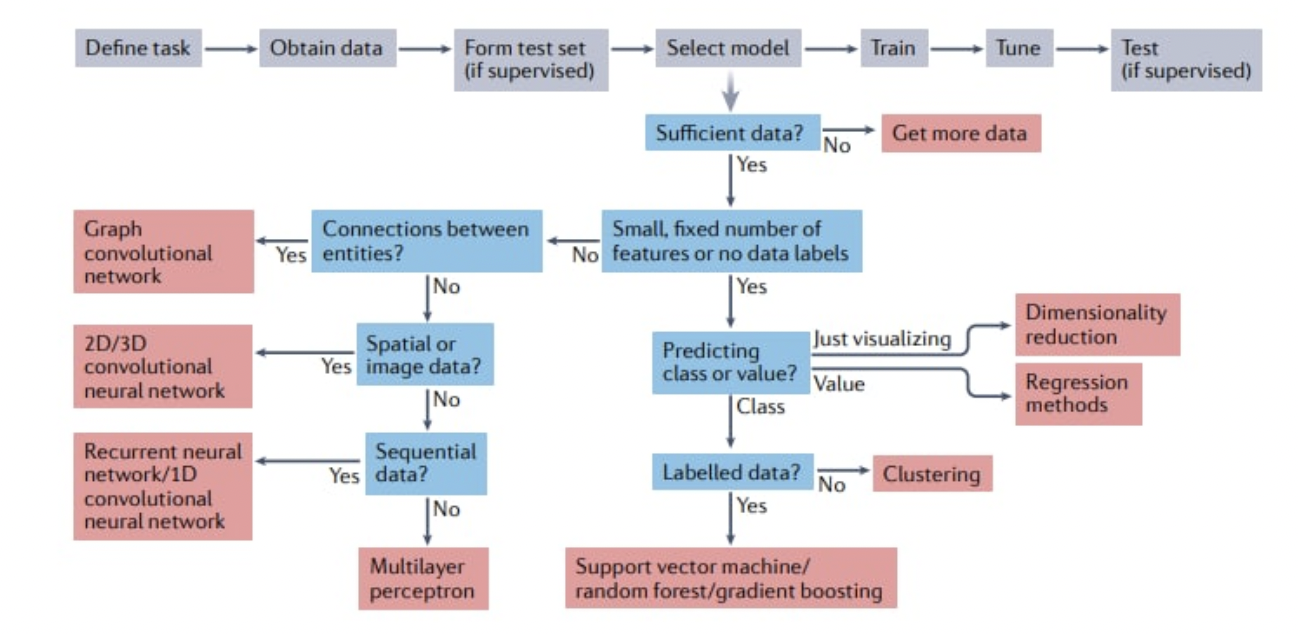
В этой же схеме присутствует фактические ошибки, например, на табличных данных нейронные сети используют редко. Как раз упомянутые на схеме бустинги (boosting) на практике используют намного чаще. Хотя и есть работы, где нейронные сети пытаются побеждать на табличных данных, признания в сообществе они не получили.
Что имеется авторами в виду под Regression methods - тоже вопрос. Куда при работе с Sequential data делись трансформеры, которые для многих задач этой области являются State of the art (SOTA), также понять сложно. Почему всё многообразие применений методов понижения размерности сведено к “просто визуализации”... Понять можно, если предположить, что авторы знали только о UMAP и tSNE. Забыв даже про PCA, изобретенный в 1901.
Еще одна проблема всей схемы, представленной авторами, — это попытка сделать ее в виде очевидного простого workflow. В результате, то же уменьшение размерности и прочее применяются только для одного кейса, хотя они могут быть частями сложных пайплайнов.
Все это приводит к тому, что по данной схеме ни один новичок вообще не поймет, как решать его задачу. А если поймет — то поймет неверно. Ведь непонятно, что такое “small … number of features”. Особенно смешно выглядит совет вида “если данных недостаточно, то наберите еще”. И в смысле “что такое недостаточно”, и в контексте современных методов, многие из которых помогают решать проблему с недостатком данных для самостоятельного обучения модели. В общем и в “педагогическом” смысле это провал.
Если идти дальше по тексту обзора, то лучше не становится. В тексте есть фактические ошибки.
Например, утверждается, что градиентный бустинг плохо подходит для задач регрессии. Тем временем, на табличных данных градиентный бустинг и для регрессии является SOTA-методом. Единственное — деревья не умеют экстраполировать. Но методов, умеющих хорошо экстраполировать данные, в общем случае в принципе нет (можно прочитать про такое явление, как covariate shift, к примеру).
А в таблице 1 (называется “Comparison of different machine learning methods”) особенно странно то, что у случайного леса и бустинга, по мнению авторов, совсем разные примеры применения. Разумеется, это не так, и обычно на задаче, где можно применить случайный лес, можно и бустинг.
Таблица 2 (называется “Recommendations for the use of machine learning strategies for different biological data types”) тоже бессмысленная. К примеру, использовать CNN для данных по экспрессии — очень нетривиальная задача. Тут как раз модель бустинга, вполне возможно, будет непобедима.
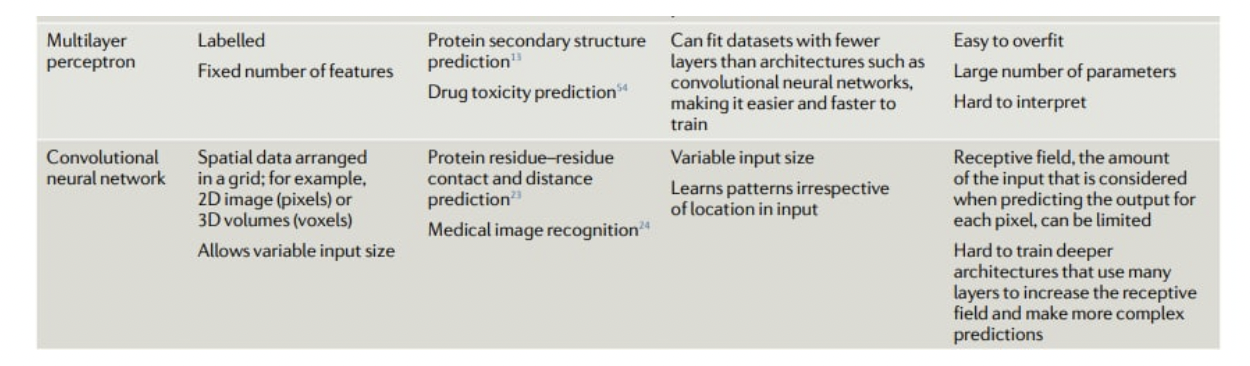
А вот в таблице (см. ниже) особенно странная фраза про то, что глубокие CNN трудно учить. Можно посмотреть на широко используемые архитектуры для работы с изображениями и увидеть, что используются ОЧЕНЬ глубокие сети — классический “стартовый” ResNet, в который можно уместить и 100+ слоев, современные SOTA — EfficientNetV2 и ConvNeXt. Всё потому, что задача с обучением глубоких сетей более-менее решена при помощи современных дополнительных слоев (например, BatchNorm) и особенностей архитектуры (residual connections).
Когда авторы говорят о data leakage, создает впечатление, что они не совсем понимают суть проблемы. И приводят не всегда валидные примеры. Что самое неприятное — есть статьи, где то, что они пытаются рассказать, рассказано подробно и ясно.
Также авторы очень много пишут про такую задачу/и, как interpretability+explainability модели. Действительно, получить в биологии (особенно в медицине) модель, которая работает средне, но при этом объясняет все свои предсказания, порой более полезно, чем получить лучшую модель, но предсказания которой абсолютно необъяснимы. Однако авторы заявляют, что классические методы по большей части легко интерпретируются, а с нейронными сетями все намного хуже.
Это не совсем так.
Проблемы с тем, чтобы на основе модели отобрать реально влияющие на что-либо признаки, могут начаться еще на этапе простой линейной модели. Спросите у экономистов.
Если мы говорим про классические модели на основе деревьев решений, то я навскидку вспоминаю 5 способов оценить вклад признака (gini, accuracy decrease, permutation importance, drop variable importance, SHAP), КАЖДЫЙ из которых обладает проблемами, подробно описанными в статьях и учебниках. И каждый из способов может дать отличный от других ответ, и каждый раз к интерпретации выхода этого способа надо прикладывать мозг. И статистическую значимость полученных результатов надо считать. А еще некоторые из этих 5 способов можно делать по-разному.
Да, если мы говорим про нейронные сети, то у них проблем с интерпретацией предсказаний минимум не меньше, а часто больше. Упомянутый SHAP есть и для нейронных сетей, и считается в имплементации автора вроде бы (автор постоянно что-то дописывает в пакете и не все отражает в документации) тремя немного разными способами. И да, на каждый способ есть своя критика. Как и на SHAP в целом.
Еще один серьезный недуг обзора — это то, что он, еще не успев выйти, успел устареть. Давайте я приведу примеры.
Очень мало сказано про трансформеры, которые во многих областях потеснили те же RNN. И начали свое восхождение трансформеры в 2017 году. В биологии их начали пытаться применять более-менее успешно почти в то же время.
Авторы зачем-то противопоставляют трансформеры и alignment-based методы. Это очень странно. Прогремевший на весь мир AlphaFold2 таки использует выравнивания. И это важная часть его пайплайна. Даже если предположить, что на момент публикации архитектура AlphaFold2 была известна недолго, то еще в феврале 2021 был опубликован MSA Transformer, который тоже работал с выравниваниями.
Почему об этом не рассказано? Мне ответ неизвестен.
Я уж молчу о том, что как раз на попытке делать выравнивание последовательностей дифференцируемым способом сосредоточено много усилий. Тенденция была видна в момент написания обзора, сейчас же есть LMA, который эту задачу решает.
Про задачу generative modelling также сказано крайне мало (есть одна ссылка по сути на статью Insilico Medicine, где именно это делают, но не так, как описали авторы обзора). Ничего не сказано про те же GAN. Информация про графовые нейронные сети, про AE рассказано куце и так, как описано, их использовать в биологии практически нельзя.
Мало сказано про такую вещь как representation learning. И, в частности, self-supervised learning. ProtTrans, DNABERT и прочее — многие современные подходы это используют. Но авторы про это не упоминают. Не рассказывают и про semi-supervised learning, который биологам в части задач может помочь. Упомянут он в одном предложении без ссылок на примеры применения.
Нет даже повсеместно использующегося transfer learning и тесно связанного fine-tuning. А ведь это — именно одно из решений для ситуации, когда у исследователя не хватает данных для тренировки своей модели с нуля. Возьми нейросеть, обученную под другую задачу, чуть поправь архитектуру, дообучи - успех. Уже упомянутые self-supervised подходы соревнуются в том, какой из них выучит представление, позволяющее лучше и с меньшим набором данных перенести модель на новую задачу.
Я перечислил, разумеется, не все минусы обзора. К сожалению, во многом этот обзор проходит по категории — легче выкинуть весь текст, поменять почти весь план статьи, сесть и написать с нуля. Чем переписывать или комментировать все ляпы, фактические и педагогические, допущенные в нем.
Такого уровня обзор можно ожидать в виде литобзора курсовой от студента ФББ МГУ старших курсов. Но как guide для кого бы то ни было, тем более, опубликованный в Nature, это не подходит ни в коем случае.
Что вы можете посоветовать на эту тему как guide для биологии?
Я не думаю, что в принципе существует такой подход — мы вырежем всё нам нужное из машинного обучения, вставим в биологию, расскажем на 14 страницах, читатель будет без проблем пользоваться полученным гомункулом. Потому идея таких гайдов “все обо всем”, возможно, в принципе обречена. Об одной только проблеме правильного разбиения данных в биологии можно написать обзорную статью того же объема, быть может, уложившись-таки в 14 страниц. Опять же, не утрирую — вот свежая статья только о том, как с медицинскими изображениями работать.
Хорошо, тогда что можно посоветовать для тех, кому интересно машинное обучение и кто хочет его изучать по хорошим, проверенным источникам?
Для тех, кто хочет заняться машинным обучением я бы советовал почитать Elements of statistical learning (Hastie et al) как введение в машинное обучение в целом.
Также может быть полезно повторить теорвер (очень классный курс) и статистику - (ссылка на курс). Без них может быть очень сложно даже на абстрактном уровне. И, понятно, нужно знание матанализа/линала хотя бы в зачаточном виде (что такое градиент, как работать с векторами и матрицами).
Кроме этого еще есть книга от Yaser Abu-Mostafa и коллег и курс от него же. На мой взгляд, второй курс сложнее.
Для введения в нейронные сети подойдет Deep Learning (Goodfellow et al). По трансформерам можно посмотреть например, вот здесь.
И лучше где-то с этого момента (не раньше, упаси вас, запускать модели, не зная, что такое переобучение) начинать гуглить статьи по конкретно своей биологической проблеме, разбираясь в деталях использованных методов.
Могу посоветовать и наш курс, который мы с коллегами читаем в МГУ при поддержке фонда Интеллект. Записи курса, пока неполные из-за проблем с несколькими лекциями, здесь. В нём мы рассказываем сначала базу, а лишь затем даём конкретные примеры из биологии. В следующем году тоже будем читать, добавив новинки и поправив недочёты.
И, на мой взгляд, есть три хороших совета, которые авторы в статье не сказали:
а) Проверьте, что ваша задача в принципе решаема. Бывают задачи, которые решить нельзя. Вообще. Доказывается математически.
b) Оцените роль случайности в вашем процессе. Какая бы не была крутая модель — для монетки она может оценить только вероятность выпадения орла/решки, а не в точности предсказать каждый исход. Потому для части задач добиться качества выше некоего порога в принципе нельзя.
c) Всегда пробуйте простые модели и “тупые” алгоритмы для решения задачи, прежде чем учить с нуля нейросеть с миллионами параметров. Иногда задачу решает просто разделение по порогу, который вы поставили, глядя на картинку. Или подход не из общего тренда машинного обучения, а именно из вашей области, который всё сделает быстро и легко. И всегда надо тестить именно такие простые модели, а потом уже уходить в дебри. В худшем случае вы просто получите базовые модели, с которыми можно сравнивать уже более сложные.
Вопросы задавала Надежда Потапова.